La plateforme SINBIOS offre aux unités en biologie et santé de la place Lilloise son expertise dans le management des données de recherche. D’une part, sur une activité de conception et d’exploitation de solutions logicielles et matérielles, pour améliorer l’acquisition, le stockage, le partage et la protection des données de recherche. D’autre part sur une activité d’intégration logicielle pour répondre à des besoins en traitement et analyse des données de recherche.
Les ingénieurs de la plateforme peuvent vous accompagner sur les problématiques de travail collaboratif et de data management dans vos projets de recherche (étude, conception, mise en production, maintenance). Chaque projet est réalisé sur mesure, en fonction de vos besoins. Nous sommes ouverts à toutes collaborations, académiques ou industrielles.
La plateforme SINBIOS est pleinement engagée dans une démarche de sobriété numérique. Ses ingénieurs sont sensibilisés et formés aux principes du numérique responsable et veillent à les appliquer au quotidien. Chaque projet intègre ainsi un volet éco-responsable, visant à réduire l’empreinte environnementale des usages numériques.
 L'actualité de la plateforme
L'actualité de la plateforme
24/10/2025 - Dans le cadre de l’appel à projets 2025 Equipements en soutien aux plateformes labellisées publié par l'Université de Lille, SINBIOS porte le projet de développement d'une solution mutualisée pour le stockage et l'analyse de données en bio-imagerie. Ce projet est co-porté par le BioImaging Center Lille (BICeL) et la plateforme ARIADNE-Criblage.
21/6/2025 - SINBIOS démarre une nouvelle collaboration avec la plateforme Bilille pour déployer une machine de calcul dédiée à l'expérience MassiveFold.
25/2/2025 - SINBIOS démarre une nouvelle collaboration avec France-BioImaging pour le déploiement d'une instance OMERO régionale. Stay tuned !
Nos compétences
Gestion de la donnée
Nous disposons d'une grande expertise dans la gestion des données de recherche, que ce soit d'un point de vue technique ou organisationnel.
Travail collaboratif
Nous proposons une solution bureautique entièrement paramétrable qui facilitera le travail collaboratif au sein vos projets de recherche.
Intégration logicielle
Nous possédons une expertise en système, réseau et virtualisation, pour intégrer des suites logicielles pour vos activités de recherche.
Conseil
Nous pouvons vous conseiller pour choisir la solution informatique la plus adaptée à votre projet de recherche.
Formation
Nous pouvons former vos équipes à l'installation et la configuration de logiciels scientifiques sur les systèmes Linux.
Sécurité
Nous pouvons vous accompagner sur des aspects techniques et organisationnels pour renforcer la sécurité de vos données de recherche.
L'équipe de la plateforme
 Intégration logicielle
Intégration logicielle
OMERO : une solution intégrée pour la gestion d'image en microscopie
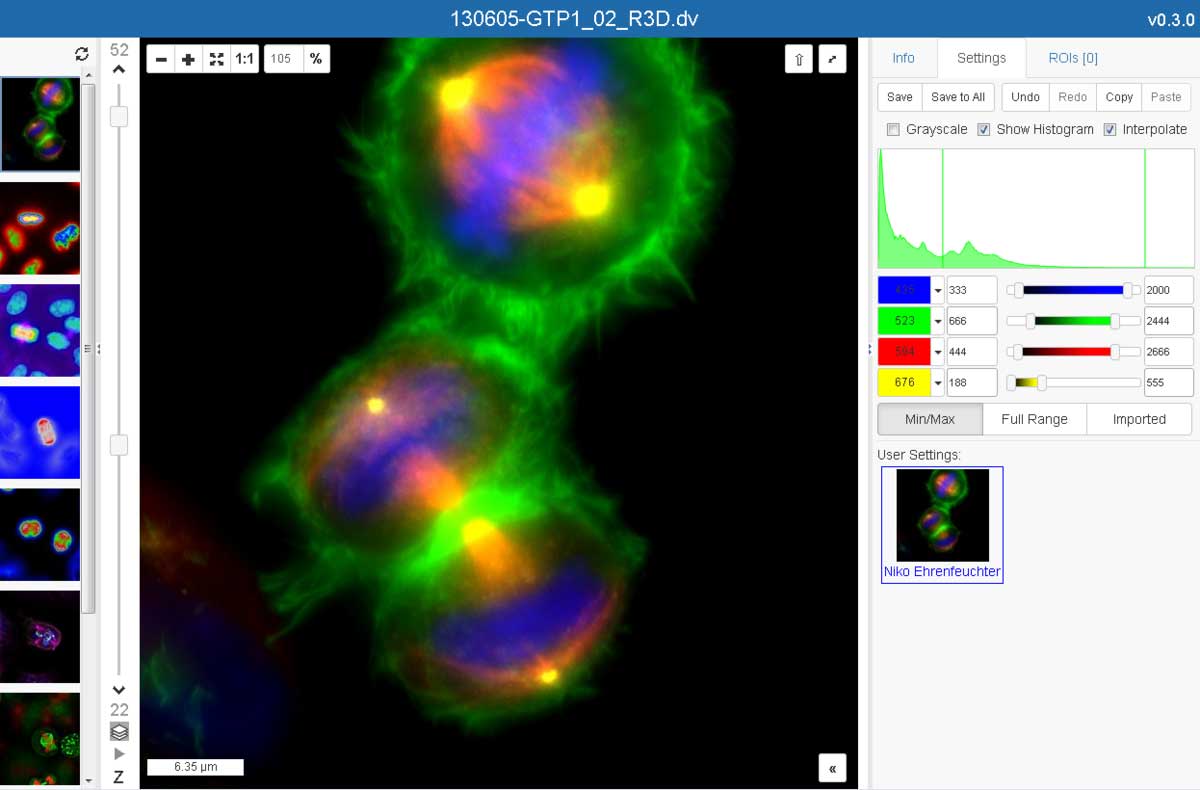
OMERO est une solution logicielle de type client-serveur permettant la visualisation, la gestion et l’analyse d’images de microscopie et les méta données associées. Plus de 140 formats de fichiers d’images sont pris en charge, y compris tous les principaux formats des microscopes commerciaux. C’est une solution full-web, qui dispose de fonctionnalités de partage et de visualisation. Elle répond parfaitement aux besoins de FAIRisation des données d'imagerie. Nous avons déployé cette solution en 2021 pour le BioImaging Center Lille (BICeL). La solution héberge aujourd'hui plusieurs milliers d'images.
 Travail collaboratif
Travail collaboratif
Nextcloud / OnlyOffice / OpenProject
OnlyOffice est une suite collaborative en ligne permettant de produire, de modifier et partager des documents bureautiques compatibles au format de fichiers Microsoft. Tous les documents sont stockés sur un espace sécurisé géré par nos soins. Nous avons intégré cette suite à la solution de partage de fichiers NextCloud pour offrir aux porteurs de projets, une solution clé en main pour la gestion des données de leurs projets de recherche. Elle permet à l’ensemble des collaborateurs d’un projet d’accéder facilement aux données. C’est une solution que nous développons sur mesure, à votre usage unique, en adéquation avec vos besoins.
Afin de proposer une solution parfaitement adaptée, nous prenons le temps d’échanger avec chaque porteur de projet pour analyser ses besoins précis. Cette approche nous permet d’ajuster finement la configuration des espaces de travail et d’optimiser leur ergonomie.
Nous offrons également la possibilité de tester une maquette très rapidement, permettant aux équipes de se familiariser avec l’outil et de valider son adéquation avant un déploiement définitif.
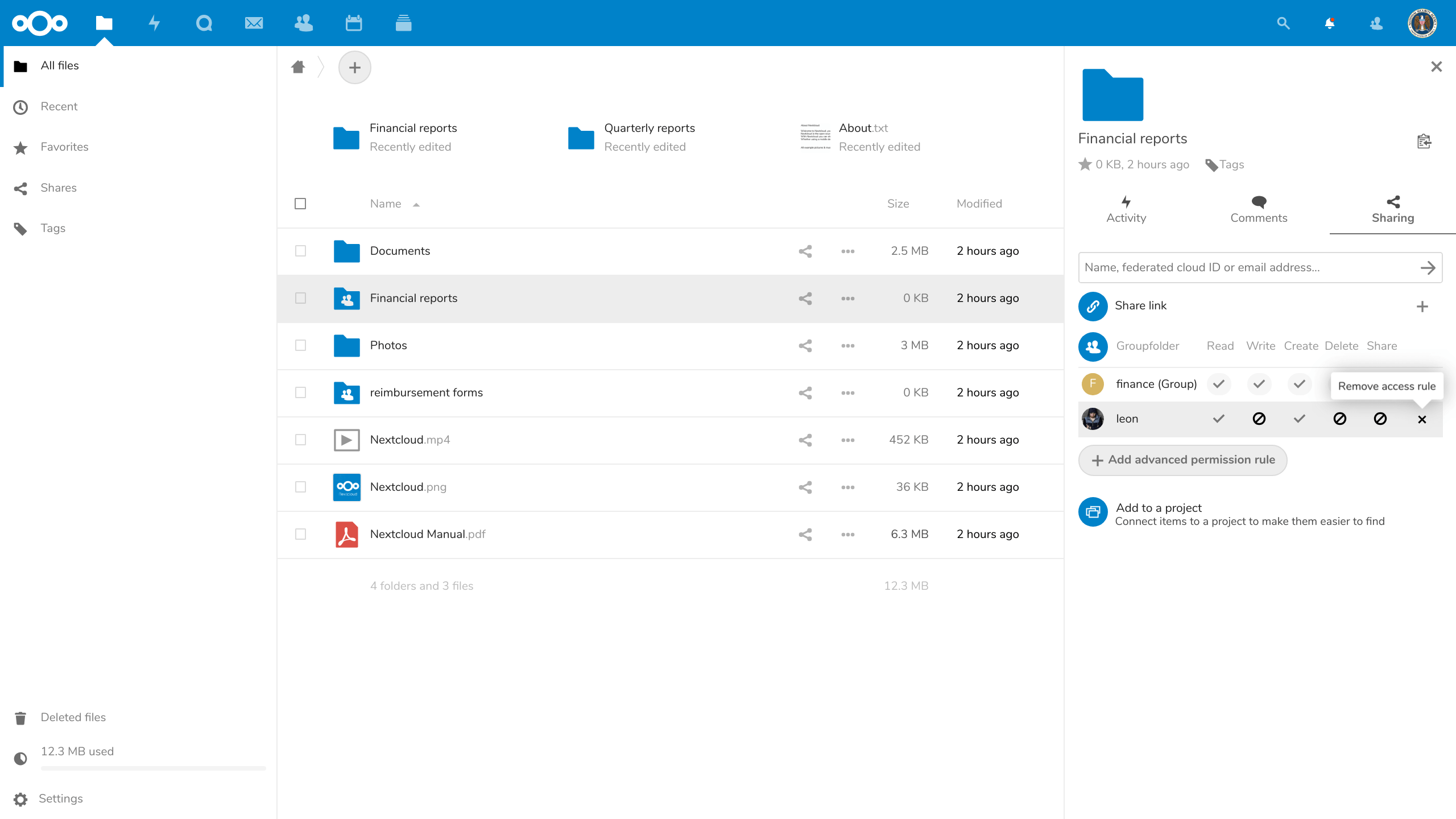
Notre engagement va au-delà de la mise en place technique : nous assurons un suivi régulier et restons disponibles pour accompagner les utilisateurs, ajuster les configurations et faire évoluer la solution en fonction des besoins.
Avec SINBIOS, chercheurs et porteurs de projet disposent d’un environnement de travail collaboratif performant, sécurisé et entièrement adapté, leur permettant de se concentrer pleinement sur leurs recherches.
Les porteurs des projets ANR Mustart et Restoltb, du projet Européén IMI Horizon 2020 ERA4TB ainsi que les organisateurs de la compétition CASP-CAPRI nous ont fait confiance.
 Intégration logicielle
Intégration logicielle
Déconvolution d'images de microscopie optique
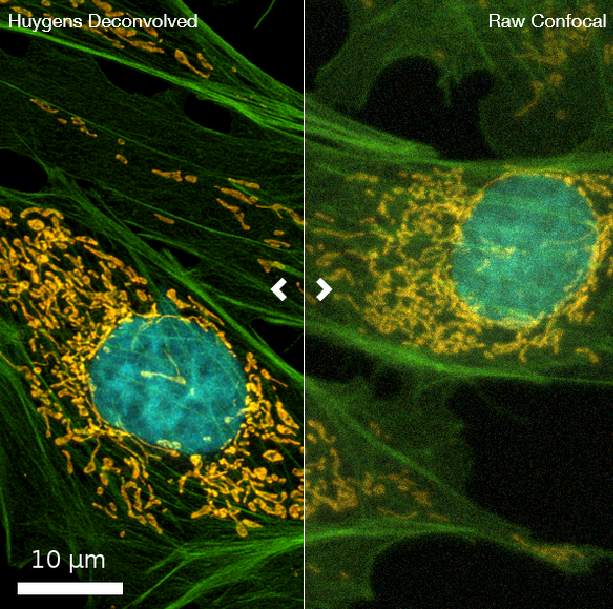
Huygens Core est une suite logicielle de traitement d'images conçue pour la restauration, la visualisation et l'analyse d'images microscopiques. A travers une interface Web, Huygens Core est capable de déconvoluer une grande variété d'images allant des images 2D à champ large aux images séries temporelles confocales multi-canaux multi-photons 4D. SINBIOS a intégré cette suite logicielle pour le BioImaging Center Lille (BICeL). Nous avons également déployé le module Remote Manager interconnecté au serveur OMERO de la plateforme pour permettre aux utilisateurs de traiter simplement des images par lots, depuis leur navigateur Web.
 Gestion des données de recherche
Gestion des données de recherche
Stockage, partage et sauvegarde
SINBIOS dispose d’une infrastructure sécurisée pour le stockage de vos données de recherche. Ce service s’adresse en priorité aux équipes ou aux porteurs de projet ayant un besoin de stockage pour une durée limitée (le temps d’un projet ou d’une expérience par exemple). Il n’a pas vocation à se substituer aux espaces de stockage mis à disposition par les tutelles, les DSI ou les grands centres de données.
Nous garantissons la disponibilité, l’accessibilité, l’intégrité et la sécurisation des données. Nous travaillons en partenariat avec les acteurs de la région pour bénéficier d’infrastructures techniques performantes. Les données sont répliquées et sauvegardées sur plusieurs sites. Elles sont accessibles via différents protocoles sécurisées.
SINBIOS peut également vous accompagner dans le choix des technologies de stockage et de sauvegarde de vos données, que ce soit sur vos propres matériels ou sur des infrastructures tierces. Vous bénéficiez ainsi de l’expertise des ingénieurs de SINBIOS pour évaluer les coûts d’investissement et de fonctionnement, mais aussi vous garantir que la solution retenue soit en parfaite adéquation avec vos besoins.
Plusieurs plateformes et unités de recherche de la région lilloise font confiance à SINBIOS pour la gestion de leurs données de recherche. Pourquoi pas vous ?
 Intégration logicielle
Intégration logicielle
Intelligence artificielle et analyse d’images en pathologie rénale
DSA est une plateforme web pour l'analyse, la visualisation, la gestion et l'annotation de données d'imagerie de pathologie numérique sur lames entières. Elle permet de stocker, de gérer, de visualiser et d'annoter de grands ensembles de données d'imagerie. C’est une plateforme qui, à travers des technologies de conteneurisation avancées, se compose d'une boîte à outils d'analyse (HistomicsTK), d'une interface pour visualiser les diapositives et gérer les annotations (HistomicsUI), d'une couche de base de données (utilisant Mongo) et d'un serveur web qui fournit une API riche et des outils de gestion des données (utilisant Girder).
Nous avons été sollicités pour collaboration dans le cadre d'un appel à projet "Jeune Chercheur" du CHU de Lille. Nous avons collaboré avec le porteur de projet pour déterminer les prérequis techniques d’une station d’analyse équipée de CPU puissants et de GPU afin de maximiser les performances tout en restant dans le budget alloué à ce projet.
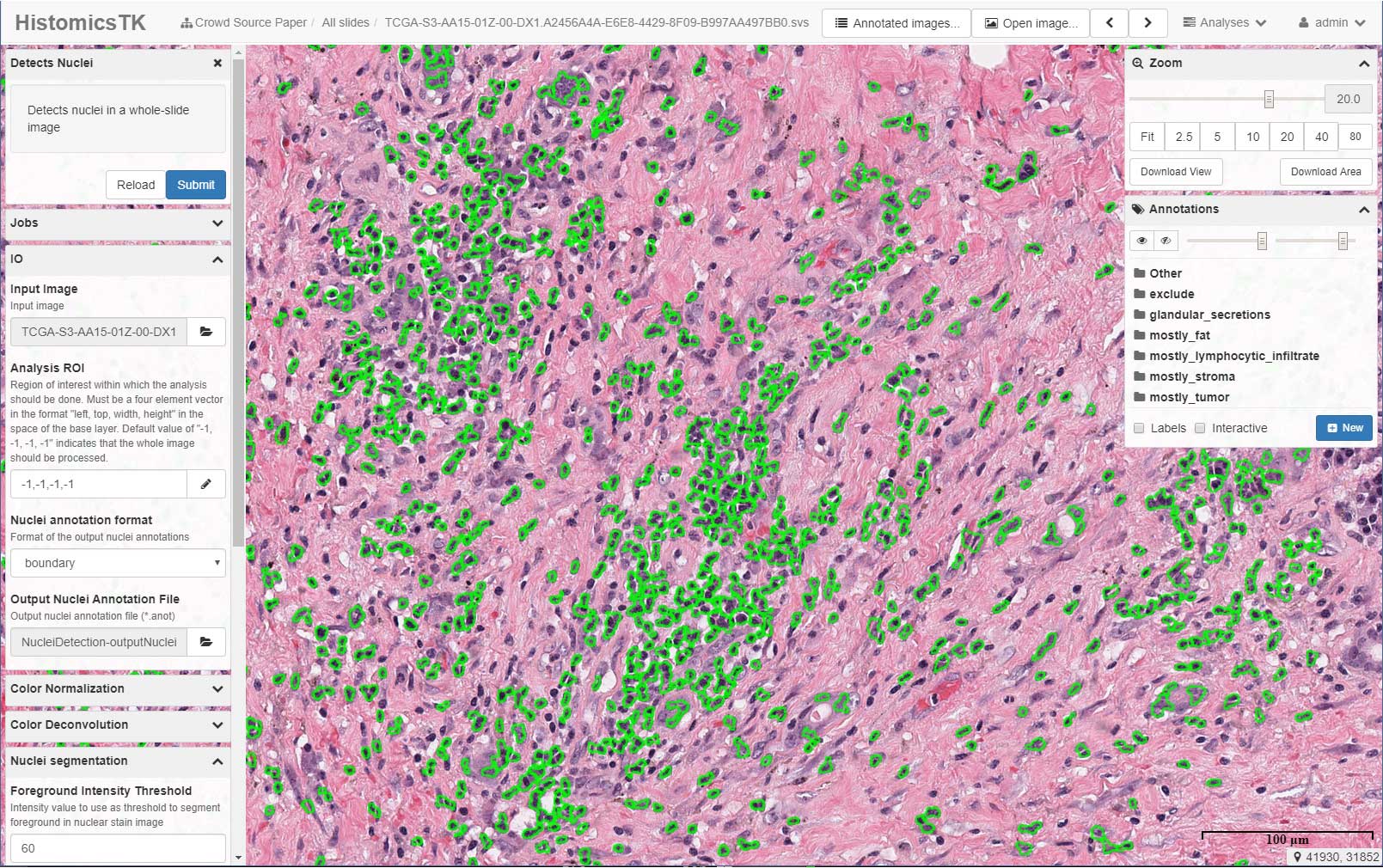
Nous avons ensuite apporté notre expertise dans l’installation et la configuration du système afin de rendre interopérables plus de quinze conteneurs Docker nécessaires au bon fonctionnement de la solution. Nous avons également optimisé certains conteneurs afin de tirer le maximum de puissance des GPU.
A la demande du porteur de projet, nous avons ajouté un serveur Orthanc sur cette station. Orthanc est un logiciel libre et open-source conçu pour gérer et stocker des images médicales, en particulier celles au format DICOM (Digital Imaging and Communications in Medicine). Il sert principalement de serveur PACS (Picture Archiving and Communication System) léger et modulaire, permettant aux établissements de santé de gérer leurs données d'imagerie médicale.
 Gestion des données de recherche
Gestion des données de recherche
Filesender par SINBIOS
Lors du premier appel à soutien lancé en 2024, SINBIOS a été sollicité par la plateforme GO@L (Génomique @ Lille) pour mettre en place une solution sécurisée d’échange de fichiers entre l’Institut Pasteur de Lille et le CHU. Cette plateforme génère des jeux de données de séquençage pouvant atteindre jusqu’à 1 téraoctet. Cependant, en raison des mesures de sécurité informatique mises en place sur les différents sites, l’utilisation de protocoles traditionnels comme FTP ou SFTP était impossible.
Après une analyse des contraintes techniques du projet – accessibilité, sécurité et flux réseau –, les ingénieurs de SINBIOS ont proposé la mise en place d’une instance dédiée de FileSender. Cette solution, spécialement configurée pour la plateforme GO@L, permet l’envoi de fichiers sans limite de taille et n’est accessible que depuis les sites concernés.
Interconnectée avec son propre annuaire, cette instance de FileSender offre également la possibilité aux administrateurs de créer des comptes personnels pour les utilisateurs non référencés dans la fédération d’identité Renater, garantissant ainsi un accès sécurisé et maîtrisé aux échanges de données.
L’équipe travaille maintenant à la mise à disposition d’une API Rest permettant de déposer des fichiers via des scripts python.
Depuis janvier 2025, SINBIOS propose désormais cette solution dans son offre de service, toujours sur la base d’une instance dédiée.





